dPCR的发展历史、原理及其应用-985毕业论文网
来源:985论文网 添加时间:2019-12-20 14:34
摘要:数字PCR(Digital PCR,dPCR)是核酸绝对定量的新方法,该技术通过分液,将含有核酸模板的PCR反应体系分配到上万个反应器中进行PCR扩增,根据荧光信号的有或无,进行结果计数,通过泊松分布的统计处理,直接得出核酸的拷贝数。相比于实时荧光定量PCR(Quantitative PCR,qPCR),dPCR不需要建立标准曲线,应用前景更广。本文对dPCR的发展历史、原理及其应用进行了综述。
关键词:数字PCR;绝对定量;拷贝数;实时荧光定量PCR.
自1985年Kary Mullis发明PCR技术以来,PCR技术一直是生物医学领域中重要的实验方法。随着分子生物学技术的发展,目前核酸定量的主要方法 是 实 时 荧 光 定 量PCR(Quantitative PCR,qPCR)。数字PCR(Digital PCR,dPCR)是近年来发展起来的新技术,是对传统PCR方法的技术革新,两者最主要的区别在于计算核酸拷贝数方法不同,dPCR方法采用了新的定量策略和实验思路,可以对核酸的拷贝数进行绝对定量,相比于qPCR不需要建立标准曲线,具有更广阔的应用前景。目前,已有很多基于微滴或芯片的商业化dPCR平台应用于分子生物学、医学等各个领域,并发挥了重要作用。因此,本文主要从dPCR方法的发展历史、原理及其应用方面进行了阐述。
1 dPCR的发展历史。
1992年,Sykes等[1]从非淋巴细胞和正常体细胞背景中鉴定突变的白血病细胞,检测了复杂背景下低丰度的IgH重链突变基因。该研究中提出了三个重要的原则:有限稀释、终点信号的有或无及对数据泊松分布的统计处理,为以后dPCR的发展奠定了基础。
1997年,有研究利用玻璃毛细管作为载体进行PCR反应,可以对模板分子进行定量,验证了Taq-Man探针可以检测模板单分子,尤其在微小的反应体系中,发展了单分子定量技术[2],为dPCR单模板扩增提供了技术支持。
1999年,Bert Vogelstein和Kenneth W.Kin-zler[3]正式提出了dPCR的概念,可以将指数型函数转化成线性函数,对模板进行有限稀释,根据荧光信号的有或无进行精确定量。实验利用384孔板对样品进行稀释、扩增,来检测结、直肠癌粪便样品中c-Ki-Ras基因突变,对数据进行泊松分布的处理。研究还提出了数字PCR的另外一个优势,由于粪便样本中含有大量的PCR反应抑制剂,通过对样品进行稀释,提高了对反应抑制剂的耐受程度。
2003年,Devin等[4]提出了BEAMing技术,包括小珠(Bead)、乳浊液(Emulsion)、扩增(Amplifi-cation)、磁性(Magnetic)。利用包被链霉亲和素的磁性珠子和生物素标记的寡核苷酸,形成微乳液,进行PCR扩增,通过流式细胞仪检测荧光标记来进行计数,这个技术为以后微滴式dPCR的产生提供了技术依据。
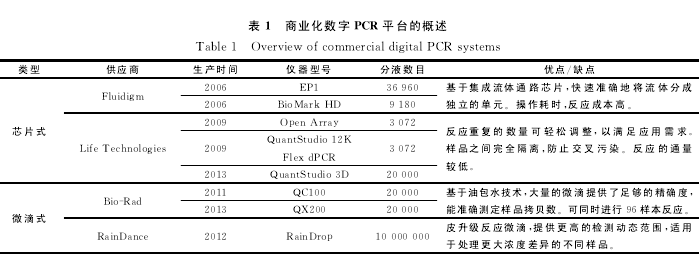
2006年,Fluidigm公司第一个生产商业化的基于芯片的dPCR仪,主要包括EP1系统和BioMarkHD系统。2009年,Life Technologies推出了OpenArray和QuantStudio 12K Flex dPCR系统,2013年,Life Technologies又 推 出 了QuantStudio 3DdPCR系统,采用高密度的纳升流控芯片技术,将样本均匀的分配至20 000个单独的反应孔中。
2011年,Bio-Rad公 司 推 出 了 基 于 微 滴 的QX100dPCR仪,利用油包水技术,将样品平均分配到20 000个微滴油包水中,利用微滴分析仪对微滴进行分析;2013年,Bio-Rad公司推出在QX100基础上的升级QX200dPCR仪。2012年RainDance公司推出了RainDrop dPCR仪,在高压气体驱动下,将每个标准反应体系分割成包含100万至1 000万个皮升级别微滴的反应乳液,提高了dPCR仪的检测范围,适于检测浓度差异较大的样品(表1)。
 到目前为止,dPCR的扩增载体从多孔板、毛细管发展到目前的芯片和微滴,降低了反应成本,提高了PCR反应的分液数目和实验的灵敏度。
到目前为止,dPCR的扩增载体从多孔板、毛细管发展到目前的芯片和微滴,降低了反应成本,提高了PCR反应的分液数目和实验的灵敏度。
2 dPCR的原理。
dPCR的原理是将含有核酸模板的标准PCR反应体系,平均分配成上万个和数百万个PCR反应,分配到芯片或微滴中,使每个反应中尽可能含有一个模板分子,进行单分子模板PCR反应,通过读取荧光信号的有或无进行计数,经过统计学泊松分布的校准进行绝对定量。
dPCR可以对样品进行绝对定量,直接检测出样品中的拷贝数,相对于qPCR,不需要建立标准曲线,灵敏度更高;dPCR通过分液,提高了PCR反应对抑制剂如SDS、EDTA、肝素的耐受程度,特别是环境、粪便样本[5];然而,qPCR由于受到抑制剂的影响,扩增效率往往会偏低(表2)。
 qPCR方法是对反应体系中荧光信号进行实时收集,通过反应的循环阈值,对初始模板进行检测和定量,主要应用于病原体的检测和定量,基因表达的相对定量,单核苷酸多态性分析,检测的动态范围广;而dPCR方法是将PCR反应体系进行分液,通过读取荧光信号的有或无进行计数,直接得出样品的拷贝数,可应用于病毒载量的绝对定量,拷贝数的变异分析,罕见突变的检测等,相比于qPCR,检测的灵敏度更高,本文也对dPCR和qPCR在应用方向上进行了比较。
qPCR方法是对反应体系中荧光信号进行实时收集,通过反应的循环阈值,对初始模板进行检测和定量,主要应用于病原体的检测和定量,基因表达的相对定量,单核苷酸多态性分析,检测的动态范围广;而dPCR方法是将PCR反应体系进行分液,通过读取荧光信号的有或无进行计数,直接得出样品的拷贝数,可应用于病毒载量的绝对定量,拷贝数的变异分析,罕见突变的检测等,相比于qPCR,检测的灵敏度更高,本文也对dPCR和qPCR在应用方向上进行了比较。
3 dPCR的应用。
3.1病毒载量的绝对定量。
目前,有 研 究 比 较 了 不 同 的dPCR平 台 和qPCR对HIV DNA的定量,结果显示QX100和Quantstudio dPCR的 变 异 系 数 分 别 为14.3%和17.5%低于qPCR的42.8%,表明dPCR的定量结果更 加 准 确;针 对 病 人 样 本HIV DNA的 检 测,QX100dPCR的检出率为80%高于qPCR的检出率62%[6].另有研究利用微滴式dPCR对戊型肝炎病毒的RNA进行定量,微滴式dPCR的分析灵敏度为80(IU)/mL,微滴式dPCR和qPCR对临床样本 的 定 量 有 很 好 的 一 致 性,秩 相 关 系 数rs为0.89[7].因此,dPCR可以对核酸的病毒载量进行绝对定量,且有较高的灵敏度。
Sanders等评价了dPCR对DNA和RNA绝对定量的准确度、灵敏度和重复性[8,9],为后期的定量研究提供了基础依据。2012年,Pinheiro等[10]确定了微滴式dPCR对Lambda DNA的检测范围在37-131 000拷贝/反应,线性关系R2=0.9994,有效的对Lambda DNA进行了定量。White等[11]第一次利用dPCR对GBV-C RNA病毒进行了绝对定量,逆转录dPCR的检出限在每反应中3~10个模板分子,表明dPCR在低丰度的样本中,有更好的扩增。有研究也利用逆转录dPCR对水生RNA病毒成功的进 行 了 定 量[12].2016年,Yan等[13]对 感 染H7N9的病人连续采取十个不同治疗期的样本进行检测定量,针对qPCR检测阴性的样本,dPCR仍检测出病毒的存在,表明dPCR定量H7N9病毒载量比qPCR更灵敏。
3.2产前诊断。
传统的产前诊断方法为侵入性的如羊膜穿刺、绒毛膜取样,这存在一定风险的胎儿丢失,利用超声波或生物化学标记对样品进行筛查。非侵入产前诊断(无创产前诊断)对胎儿的影响较小,检测更加准确,成为目前新的诊断方法。1997年,Lo等[14]发现母体内胎儿游离DNA(Cell-freefetal DNA,cffDNA)的存在,为无创产前检测提供了可能。2007年,Lo等[15]利用新的dPCR方法无创伤的检测了cffDNA中的非整倍染色体,通过检测孕妇血浆中21号染色体上PLAC4 mRNA的SNP等位基因的不平衡性;及评价胎儿DNA中21号染色体的含量与参照染色体相比是否过表达,可检测含有25%cffDNA血浆样本中21号染色体的整倍性。有研究比较了dPCR和RT-PCR对孕妇血浆中cffDNA的定量效果,表明微流体数字PCR比RT-PCR有更小的偏倚,dPCR的定量准确度是传统RT-PCR的3.1倍,两者定量的变异系数分别为16%和49%,检 测 的 灵 敏 度 分 别 为100%和90%[16].2012年,Barrett等[17]利用dPCR的方法对孕妇血浆中cffDNA进行了镰状红细胞贫血的检测。此外,dPCR在检测样本中罕见基因突变上也有自己的优势,有研究利用dPCR检测了X染色体的基因位点,成功识别了血友病的突变基因及胎儿的Rh血型[18,19].
3.3癌症诊断检测。
MicroRNA(miRNA)是一类由内源基因编码的长度约为19~24个核苷酸的非编码单链RNA分子。MicroRNA在细胞增殖、分化与凋亡过程中发挥重要功能,其也与癌症的发生关系密切,被当作癌症标志物[20,21].Hindson等[22]系统的建立了微滴式dPCR检测和定量microRNA的方法。2013年,Ma等[23]利用微滴式dPCR技术定量了非小细胞性肺癌病人血浆中低丰度的mi-croRNA的含量,dPCR对microRNA的检测范围在1~10 000拷贝/μL;相比qPCR,dPCR定量mi-croRNA的拷贝数 有更高 的 灵 敏 度,可 对 血 浆 中miR-21-5p和miR-335-3p定 量,qPCR只 能 定 量miR-21-5p.通过利用QuantStudio 3DdPCR也成功对血浆样本中的microRNA的含量进行了有效的定量[24].2014年,Li等[25]利用微滴dPCR定量痰标本中的microRNA进行肺癌诊断,表明dPCR可以对痰标本中的miR-31和miR-210绝对定量,为肺癌的临床诊断提供了新的技术。有研究利用dPCR技术检测样品中极微量的基因和罕见基因突变,如表皮生长因子及K-RAS基因突变,对癌症进行诊断检测[26-28].
3.4下一代测序技术的应用。
随着测序技术的发展,下一代测序技术(Next generation sequencing,NGS)应运而生。近年来,NGS技术得到了快速的发展,特别是在临床病原微生物的鉴定,发挥了重大作用[29].为了获得高质量的测序结果,测序文库的准确定量是必要的。White等[30]利用微流体dPCR对454和Solexa测序平台的文库进行了准确定量,dPCR的 变 异 系 数 为11.8%明 显 低 于qPCR的21.2%,结果更加准确,并对纳克级的细菌及哺乳动物的DNA进行了测序。通过对NGS文库定量方法的比较,dPCR的定量结果更加灵敏和准确[31].利用dPCR方法实现了测序文库的绝对定量,降低了样本的用量。
3.5转基因成分检测。
传统的qPCR方法可以对食品中的转基因成分进行测定[32,33],由于该方法需要建立标准曲线,加上食品与作物等复杂样品中抑制剂对PCR扩增效率的影响,使qPCR对于低丰度的转基因成分的检测有很大的局限[34].有研究利用dPCR定量转基因MON810与参考基因hmg基因拷贝数的比值,定量结果与qPCR相同[35].2013年,Morisset等[36]利用微滴式dPCR双重检测转基因MON810和参考基因hmg拷贝数比值,微滴式dPCR检测hmg和MON810的线性范围分别为:5~118 000拷贝/反应和6~4 340拷贝/反应,检测的线性范围较宽,能满足常规转基因成分检测的要求。有研究报道dPCR检测转基因成分的灵敏度为0.1%,低 于 欧 盟 规 定 转 基 因 成 分 的 检 出 限0.9%[37].研究表明dPCR可以作为常规的检测技术应用于转基因成分鉴定。
4结语与展望。
dPCR作为核酸定量的新技术,与此前的核酸定量方法相比,方法的灵敏度、准确度更高。dPCR为分子生物学、微生物学等领域提供了新的检测方法和实验思路。从应用的范围、实验的成本角度来看,dPCR不可能完全取代qPCR,但是dPCR方法可以进行精准的绝对定量分析,极好的数据重现性,而且样本需求较低,在核酸检测与定量等方面有非常重要的补充。在整个dPCR的发展过程中,应用越来越广泛,在核酸检测定量、抗病毒治疗监测、预后判断具有重要的意义。随着dPCR技术和商品化平台的发展,dPCR的通量将会更高,成本更低,在科学研究领域将会发挥更重要的作用。
参考文献:
[1]Sykes P J,Neoh S H,Brisco M J,et al.Quantitation oftargets for PCR by use of limiting dilution[J].Biotech-niques,1992,13(3):444-449.
[2]Olga K,Irina L,James B,et al.Nanoliter scale PCRwith TaqMan detection[J].Nucleic Acids Res,1997,25(10):1999-2004.
[3]Vogelstein B,Kinzler K W.Digital PCR[J].PNAS,1999,96(16):9236-9241.
[4]Devin D,Hai Y,Giovanni T,et al.Transforming sin-gle DNA molecules into fluorescent magnetic particlesfor detection and enumeration of genetic variations[J].PNAS,2003,100(15):8817-8822.
[5]Dingle TC,Sedlak R H,Cook L,et al.Tolerance ofdroplet-digital PCR versus real-time quantitative PCR toinhibitory substances[J].Clin Chem,2013,59(11):1670-1672.
[6]Bosman K J,Nijhuis M,Van Ham P M,et al.Compar-ison of digital PCR platforms and semi-nested qPCR as atool to determine the size of the HIV reservoir[J].Sci-entific Reports,2015,5:13811.
[7]Nicot F,Cazabat M,Lhomme S,et al.Quantificationof HEV RNA by Droplet Digital PCR[J].Viruses,2016,8:233.
[8]Sanders R,Huggett J F,Bushell C A,et al.Evaluationof digital PCR for absolute DNA quantification[J].A-nal Chem,2011,83,6474-6484.
[9]Sanders R,Mason D J,Foy C A,et al.Evaluation ofdigital PCR for absolute RNA quantification[J/OL].PLoS One,2013,8(9):e75296.
[10]Pinheiro L B,Coleman V A,Hindson C M,et al.E-valuation of a droplet digital polymerase chain reactionformat for DNA copy number quantification[J].AnalChem,2012,84:1003-1011.
[11]White R A,Quake S R,Curr K.Digital PCR providesabsolute quantitation of viral load for an occult RNA vi-rus[J].J Virol Methods,2012,179:45-50.
[12]Racki N,Morisset D,Gutierrez A I,et al.One-stepRT-droplet digital PCR:a breakthrough in the quanti-fication of waterborne RNA viruses[J].Anal BioanalChem 2014,406:661-667.
[13]Yan Yong,Jia X J,Wang H H,et al.Dynamic quan-tification of avian influenza H7N9(A)virus in a humaninfection during clinical treatment using droplet digitalPCR[J].J Virol Methods,2016,234:22-27.
[14]Lo Y M D,Corbetta N,Chamberlain P F,et al.Pres-ence of fetal DNA in maternal plasma and serum[J].The Lancet,350(9076):485-487.
[15]Lo Y M D,Fiona M F,Chan K C A,et al.DigitalPCR for the molecular detection of fetal chromosomalaneuploidy[J].Proc Natl Acad Sci USA,2007,104(32):13116-13121.
[16]Lun F M F,Allen C K,Yeung L T,et al.Microfluid-ics digital PCR reveals a higher than expected fractionof fetal DNA in maternal plasma[J].Clin Chem,2008,54(10):1664-1672.
[17]Barrett A N,McDonnell T C,Chan K C et al.DigitalPCR analysis of maternal plasma for noninvasive detec-tion of sickle cell anemia[J].Clinical Chemistry,2012,58:6 1026-1032.
[18]Tsui N B,Kadir R A,Chan K C,et al.Noninvasiveprenatal diagnosis of hemophilia by microfluidics digitalPCR analysis of maternal plasma DNA[J].Blood,2011,117(13):3684-3691.
[19]Tsui N B,Hyland C A,Gardener G J,et al.Noninva-sive fetal RHD genotyping by microfluidics digital PCRusing maternal plasma from two alloimmunized withthe variant RHD(IVS3+1G>A)allele[J].Prenat Di-agn,2013,33(12):1214-1216.
[20]Lu J,Getz G,Miska E A,et al.MicroRNA expres-sion profiles classify human cancers.[J]Nature,2005,435(7043):834-838.
[21]Yanaihara N,Caplen N,Bowman E,et al.Unique mi-croRNA molecular profiles in lung cancerdiagnosis andprognosis[J].Cancer Cell,2006,9(3):189-198.
[22]Hindson C M,Chevillet J R,Briggs H A,et al.Abso-lute quantification by droplet digital PCR versus analogreal-time PCR[J].Nat Methods,2013,10(10):1003-1005.
[23]Ma J,Li N,Guarnera M,et al.Quantification of plas-ma miRNAs by digital PCR for cancer diagnosis[J].Biomarker Insights,2013,8:127-136.
[24]Conte D,Verri C,Borzi C et al.Novel method to de-tect microRNAs using chip-based QuantStudio 3Ddig-ital PCR[J].BMC Genomics,2015,16:849.
[25]Li N,Ma J,Guarnera M,et al.Digital PCR quantifi-cation of miRNAs in sputum for diagnosis of lung canc-er[J].J Cancer Res Clin Oncol,2014,140(1):145-150.
[26]Yung T K,Chan K A,Mok T S,et al.Sing-moleculedetection of epidermal growth factor receptor mutationsin plasma by microfluidics digital PCR in non-small celllung cancer patients[J].Clinical Cancer Research,2009,15(6):2076-2084.
[27]Wang J,Ramakrishnan R,Tang Z,et al.QuantifyingEGFR alterations in the lung cancer genome withnanofluidic digital PCR arrays[J].Clin Chem,2010,56(4):623-632.
[28]Taly V,Pekin D,Benhaim L,et al.Multiplex picodr-oplet digital PCR to detect KRAS mutation in circulat-ing DNA from the plasma of colorectal cancer patients[J].Clin Chem,2013,59(12):1722-1731.
[29]Jacob H J.Next-generation sequencing for clinical di-agnostics[J].N Engl J Med,2013,369:1557-1558.
[30]White R A,Blainey P C,Fan H C,et al.Digital PCRprovides sensitive and absolute calibration for highthroughput sequencing[J].BMC Genomics,2009,10:116.
[31]Robin J D,Ludlow A T,LaRanger R,et al.Compari-son of DNA quantification methods for Next generationsequencing[J].Scientific Reports,2016,6:24067.
[32]Holst-Jensen A.Testing for genetically modified or-ganisms(GMOs):Past,present and future perspec-tives[J].Biotechnol Adv,2009,27:1071-1082.
[33]Settanni L,Corsetti A.The use of multiplex PCR todetect and differentiate food-and beverage-associatedmicroorganisms:A review[J].Journal of Microbiolog-ical Methods,2007,69:1-22.
[34]Cankar K,Stebih D,Dreo T,et al.Critical points ofDNA quantification by real-time PCR-effects of DNAextraction method and sample matrix on quantificationof genetically modified organisms[J].BMC Biotechnol,2006,6:37.
[35]Corbisier P,Bhat S,Partis L,et al.Absolute quantifi-cation of genetically modified MON810maize by digitalpolymerase chain reaction[J].Analytical and Bioana-lytical Chenistry,2010,396(6):2143-2150.
[36]Morisset D,Stebih D,Milavec M,et al.Quantitativeanalysis of food and feed samples with droplet digitalPCR[J/OL].PLoS One,2013,8(5):e62583.
[37]Fu W,Zhu P Y,Wang C G,et al.A highly sensitiveand specific method for the screening detection of ge-netically modified organisms based on digital PCR with-out pretreatment[J].Scientific Reports,2015,5:12715
关键词:数字PCR;绝对定量;拷贝数;实时荧光定量PCR.
自1985年Kary Mullis发明PCR技术以来,PCR技术一直是生物医学领域中重要的实验方法。随着分子生物学技术的发展,目前核酸定量的主要方法 是 实 时 荧 光 定 量PCR(Quantitative PCR,qPCR)。数字PCR(Digital PCR,dPCR)是近年来发展起来的新技术,是对传统PCR方法的技术革新,两者最主要的区别在于计算核酸拷贝数方法不同,dPCR方法采用了新的定量策略和实验思路,可以对核酸的拷贝数进行绝对定量,相比于qPCR不需要建立标准曲线,具有更广阔的应用前景。目前,已有很多基于微滴或芯片的商业化dPCR平台应用于分子生物学、医学等各个领域,并发挥了重要作用。因此,本文主要从dPCR方法的发展历史、原理及其应用方面进行了阐述。
1 dPCR的发展历史。
1992年,Sykes等[1]从非淋巴细胞和正常体细胞背景中鉴定突变的白血病细胞,检测了复杂背景下低丰度的IgH重链突变基因。该研究中提出了三个重要的原则:有限稀释、终点信号的有或无及对数据泊松分布的统计处理,为以后dPCR的发展奠定了基础。
1997年,有研究利用玻璃毛细管作为载体进行PCR反应,可以对模板分子进行定量,验证了Taq-Man探针可以检测模板单分子,尤其在微小的反应体系中,发展了单分子定量技术[2],为dPCR单模板扩增提供了技术支持。
1999年,Bert Vogelstein和Kenneth W.Kin-zler[3]正式提出了dPCR的概念,可以将指数型函数转化成线性函数,对模板进行有限稀释,根据荧光信号的有或无进行精确定量。实验利用384孔板对样品进行稀释、扩增,来检测结、直肠癌粪便样品中c-Ki-Ras基因突变,对数据进行泊松分布的处理。研究还提出了数字PCR的另外一个优势,由于粪便样本中含有大量的PCR反应抑制剂,通过对样品进行稀释,提高了对反应抑制剂的耐受程度。
2003年,Devin等[4]提出了BEAMing技术,包括小珠(Bead)、乳浊液(Emulsion)、扩增(Amplifi-cation)、磁性(Magnetic)。利用包被链霉亲和素的磁性珠子和生物素标记的寡核苷酸,形成微乳液,进行PCR扩增,通过流式细胞仪检测荧光标记来进行计数,这个技术为以后微滴式dPCR的产生提供了技术依据。
2006年,Fluidigm公司第一个生产商业化的基于芯片的dPCR仪,主要包括EP1系统和BioMarkHD系统。2009年,Life Technologies推出了OpenArray和QuantStudio 12K Flex dPCR系统,2013年,Life Technologies又 推 出 了QuantStudio 3DdPCR系统,采用高密度的纳升流控芯片技术,将样本均匀的分配至20 000个单独的反应孔中。
2011年,Bio-Rad公 司 推 出 了 基 于 微 滴 的QX100dPCR仪,利用油包水技术,将样品平均分配到20 000个微滴油包水中,利用微滴分析仪对微滴进行分析;2013年,Bio-Rad公司推出在QX100基础上的升级QX200dPCR仪。2012年RainDance公司推出了RainDrop dPCR仪,在高压气体驱动下,将每个标准反应体系分割成包含100万至1 000万个皮升级别微滴的反应乳液,提高了dPCR仪的检测范围,适于检测浓度差异较大的样品(表1)。

2 dPCR的原理。
dPCR的原理是将含有核酸模板的标准PCR反应体系,平均分配成上万个和数百万个PCR反应,分配到芯片或微滴中,使每个反应中尽可能含有一个模板分子,进行单分子模板PCR反应,通过读取荧光信号的有或无进行计数,经过统计学泊松分布的校准进行绝对定量。
dPCR可以对样品进行绝对定量,直接检测出样品中的拷贝数,相对于qPCR,不需要建立标准曲线,灵敏度更高;dPCR通过分液,提高了PCR反应对抑制剂如SDS、EDTA、肝素的耐受程度,特别是环境、粪便样本[5];然而,qPCR由于受到抑制剂的影响,扩增效率往往会偏低(表2)。

3 dPCR的应用。
3.1病毒载量的绝对定量。
目前,有 研 究 比 较 了 不 同 的dPCR平 台 和qPCR对HIV DNA的定量,结果显示QX100和Quantstudio dPCR的 变 异 系 数 分 别 为14.3%和17.5%低于qPCR的42.8%,表明dPCR的定量结果更 加 准 确;针 对 病 人 样 本HIV DNA的 检 测,QX100dPCR的检出率为80%高于qPCR的检出率62%[6].另有研究利用微滴式dPCR对戊型肝炎病毒的RNA进行定量,微滴式dPCR的分析灵敏度为80(IU)/mL,微滴式dPCR和qPCR对临床样本 的 定 量 有 很 好 的 一 致 性,秩 相 关 系 数rs为0.89[7].因此,dPCR可以对核酸的病毒载量进行绝对定量,且有较高的灵敏度。
Sanders等评价了dPCR对DNA和RNA绝对定量的准确度、灵敏度和重复性[8,9],为后期的定量研究提供了基础依据。2012年,Pinheiro等[10]确定了微滴式dPCR对Lambda DNA的检测范围在37-131 000拷贝/反应,线性关系R2=0.9994,有效的对Lambda DNA进行了定量。White等[11]第一次利用dPCR对GBV-C RNA病毒进行了绝对定量,逆转录dPCR的检出限在每反应中3~10个模板分子,表明dPCR在低丰度的样本中,有更好的扩增。有研究也利用逆转录dPCR对水生RNA病毒成功的进 行 了 定 量[12].2016年,Yan等[13]对 感 染H7N9的病人连续采取十个不同治疗期的样本进行检测定量,针对qPCR检测阴性的样本,dPCR仍检测出病毒的存在,表明dPCR定量H7N9病毒载量比qPCR更灵敏。
3.2产前诊断。
传统的产前诊断方法为侵入性的如羊膜穿刺、绒毛膜取样,这存在一定风险的胎儿丢失,利用超声波或生物化学标记对样品进行筛查。非侵入产前诊断(无创产前诊断)对胎儿的影响较小,检测更加准确,成为目前新的诊断方法。1997年,Lo等[14]发现母体内胎儿游离DNA(Cell-freefetal DNA,cffDNA)的存在,为无创产前检测提供了可能。2007年,Lo等[15]利用新的dPCR方法无创伤的检测了cffDNA中的非整倍染色体,通过检测孕妇血浆中21号染色体上PLAC4 mRNA的SNP等位基因的不平衡性;及评价胎儿DNA中21号染色体的含量与参照染色体相比是否过表达,可检测含有25%cffDNA血浆样本中21号染色体的整倍性。有研究比较了dPCR和RT-PCR对孕妇血浆中cffDNA的定量效果,表明微流体数字PCR比RT-PCR有更小的偏倚,dPCR的定量准确度是传统RT-PCR的3.1倍,两者定量的变异系数分别为16%和49%,检 测 的 灵 敏 度 分 别 为100%和90%[16].2012年,Barrett等[17]利用dPCR的方法对孕妇血浆中cffDNA进行了镰状红细胞贫血的检测。此外,dPCR在检测样本中罕见基因突变上也有自己的优势,有研究利用dPCR检测了X染色体的基因位点,成功识别了血友病的突变基因及胎儿的Rh血型[18,19].
3.3癌症诊断检测。
MicroRNA(miRNA)是一类由内源基因编码的长度约为19~24个核苷酸的非编码单链RNA分子。MicroRNA在细胞增殖、分化与凋亡过程中发挥重要功能,其也与癌症的发生关系密切,被当作癌症标志物[20,21].Hindson等[22]系统的建立了微滴式dPCR检测和定量microRNA的方法。2013年,Ma等[23]利用微滴式dPCR技术定量了非小细胞性肺癌病人血浆中低丰度的mi-croRNA的含量,dPCR对microRNA的检测范围在1~10 000拷贝/μL;相比qPCR,dPCR定量mi-croRNA的拷贝数 有更高 的 灵 敏 度,可 对 血 浆 中miR-21-5p和miR-335-3p定 量,qPCR只 能 定 量miR-21-5p.通过利用QuantStudio 3DdPCR也成功对血浆样本中的microRNA的含量进行了有效的定量[24].2014年,Li等[25]利用微滴dPCR定量痰标本中的microRNA进行肺癌诊断,表明dPCR可以对痰标本中的miR-31和miR-210绝对定量,为肺癌的临床诊断提供了新的技术。有研究利用dPCR技术检测样品中极微量的基因和罕见基因突变,如表皮生长因子及K-RAS基因突变,对癌症进行诊断检测[26-28].
3.4下一代测序技术的应用。
随着测序技术的发展,下一代测序技术(Next generation sequencing,NGS)应运而生。近年来,NGS技术得到了快速的发展,特别是在临床病原微生物的鉴定,发挥了重大作用[29].为了获得高质量的测序结果,测序文库的准确定量是必要的。White等[30]利用微流体dPCR对454和Solexa测序平台的文库进行了准确定量,dPCR的 变 异 系 数 为11.8%明 显 低 于qPCR的21.2%,结果更加准确,并对纳克级的细菌及哺乳动物的DNA进行了测序。通过对NGS文库定量方法的比较,dPCR的定量结果更加灵敏和准确[31].利用dPCR方法实现了测序文库的绝对定量,降低了样本的用量。
3.5转基因成分检测。
传统的qPCR方法可以对食品中的转基因成分进行测定[32,33],由于该方法需要建立标准曲线,加上食品与作物等复杂样品中抑制剂对PCR扩增效率的影响,使qPCR对于低丰度的转基因成分的检测有很大的局限[34].有研究利用dPCR定量转基因MON810与参考基因hmg基因拷贝数的比值,定量结果与qPCR相同[35].2013年,Morisset等[36]利用微滴式dPCR双重检测转基因MON810和参考基因hmg拷贝数比值,微滴式dPCR检测hmg和MON810的线性范围分别为:5~118 000拷贝/反应和6~4 340拷贝/反应,检测的线性范围较宽,能满足常规转基因成分检测的要求。有研究报道dPCR检测转基因成分的灵敏度为0.1%,低 于 欧 盟 规 定 转 基 因 成 分 的 检 出 限0.9%[37].研究表明dPCR可以作为常规的检测技术应用于转基因成分鉴定。
4结语与展望。
dPCR作为核酸定量的新技术,与此前的核酸定量方法相比,方法的灵敏度、准确度更高。dPCR为分子生物学、微生物学等领域提供了新的检测方法和实验思路。从应用的范围、实验的成本角度来看,dPCR不可能完全取代qPCR,但是dPCR方法可以进行精准的绝对定量分析,极好的数据重现性,而且样本需求较低,在核酸检测与定量等方面有非常重要的补充。在整个dPCR的发展过程中,应用越来越广泛,在核酸检测定量、抗病毒治疗监测、预后判断具有重要的意义。随着dPCR技术和商品化平台的发展,dPCR的通量将会更高,成本更低,在科学研究领域将会发挥更重要的作用。
参考文献:
[1]Sykes P J,Neoh S H,Brisco M J,et al.Quantitation oftargets for PCR by use of limiting dilution[J].Biotech-niques,1992,13(3):444-449.
[2]Olga K,Irina L,James B,et al.Nanoliter scale PCRwith TaqMan detection[J].Nucleic Acids Res,1997,25(10):1999-2004.
[3]Vogelstein B,Kinzler K W.Digital PCR[J].PNAS,1999,96(16):9236-9241.
[4]Devin D,Hai Y,Giovanni T,et al.Transforming sin-gle DNA molecules into fluorescent magnetic particlesfor detection and enumeration of genetic variations[J].PNAS,2003,100(15):8817-8822.
[5]Dingle TC,Sedlak R H,Cook L,et al.Tolerance ofdroplet-digital PCR versus real-time quantitative PCR toinhibitory substances[J].Clin Chem,2013,59(11):1670-1672.
[6]Bosman K J,Nijhuis M,Van Ham P M,et al.Compar-ison of digital PCR platforms and semi-nested qPCR as atool to determine the size of the HIV reservoir[J].Sci-entific Reports,2015,5:13811.
[7]Nicot F,Cazabat M,Lhomme S,et al.Quantificationof HEV RNA by Droplet Digital PCR[J].Viruses,2016,8:233.
[8]Sanders R,Huggett J F,Bushell C A,et al.Evaluationof digital PCR for absolute DNA quantification[J].A-nal Chem,2011,83,6474-6484.
[9]Sanders R,Mason D J,Foy C A,et al.Evaluation ofdigital PCR for absolute RNA quantification[J/OL].PLoS One,2013,8(9):e75296.
[10]Pinheiro L B,Coleman V A,Hindson C M,et al.E-valuation of a droplet digital polymerase chain reactionformat for DNA copy number quantification[J].AnalChem,2012,84:1003-1011.
[11]White R A,Quake S R,Curr K.Digital PCR providesabsolute quantitation of viral load for an occult RNA vi-rus[J].J Virol Methods,2012,179:45-50.
[12]Racki N,Morisset D,Gutierrez A I,et al.One-stepRT-droplet digital PCR:a breakthrough in the quanti-fication of waterborne RNA viruses[J].Anal BioanalChem 2014,406:661-667.
[13]Yan Yong,Jia X J,Wang H H,et al.Dynamic quan-tification of avian influenza H7N9(A)virus in a humaninfection during clinical treatment using droplet digitalPCR[J].J Virol Methods,2016,234:22-27.
[14]Lo Y M D,Corbetta N,Chamberlain P F,et al.Pres-ence of fetal DNA in maternal plasma and serum[J].The Lancet,350(9076):485-487.
[15]Lo Y M D,Fiona M F,Chan K C A,et al.DigitalPCR for the molecular detection of fetal chromosomalaneuploidy[J].Proc Natl Acad Sci USA,2007,104(32):13116-13121.
[16]Lun F M F,Allen C K,Yeung L T,et al.Microfluid-ics digital PCR reveals a higher than expected fractionof fetal DNA in maternal plasma[J].Clin Chem,2008,54(10):1664-1672.
[17]Barrett A N,McDonnell T C,Chan K C et al.DigitalPCR analysis of maternal plasma for noninvasive detec-tion of sickle cell anemia[J].Clinical Chemistry,2012,58:6 1026-1032.
[18]Tsui N B,Kadir R A,Chan K C,et al.Noninvasiveprenatal diagnosis of hemophilia by microfluidics digitalPCR analysis of maternal plasma DNA[J].Blood,2011,117(13):3684-3691.
[19]Tsui N B,Hyland C A,Gardener G J,et al.Noninva-sive fetal RHD genotyping by microfluidics digital PCRusing maternal plasma from two alloimmunized withthe variant RHD(IVS3+1G>A)allele[J].Prenat Di-agn,2013,33(12):1214-1216.
[20]Lu J,Getz G,Miska E A,et al.MicroRNA expres-sion profiles classify human cancers.[J]Nature,2005,435(7043):834-838.
[21]Yanaihara N,Caplen N,Bowman E,et al.Unique mi-croRNA molecular profiles in lung cancerdiagnosis andprognosis[J].Cancer Cell,2006,9(3):189-198.
[22]Hindson C M,Chevillet J R,Briggs H A,et al.Abso-lute quantification by droplet digital PCR versus analogreal-time PCR[J].Nat Methods,2013,10(10):1003-1005.
[23]Ma J,Li N,Guarnera M,et al.Quantification of plas-ma miRNAs by digital PCR for cancer diagnosis[J].Biomarker Insights,2013,8:127-136.
[24]Conte D,Verri C,Borzi C et al.Novel method to de-tect microRNAs using chip-based QuantStudio 3Ddig-ital PCR[J].BMC Genomics,2015,16:849.
[25]Li N,Ma J,Guarnera M,et al.Digital PCR quantifi-cation of miRNAs in sputum for diagnosis of lung canc-er[J].J Cancer Res Clin Oncol,2014,140(1):145-150.
[26]Yung T K,Chan K A,Mok T S,et al.Sing-moleculedetection of epidermal growth factor receptor mutationsin plasma by microfluidics digital PCR in non-small celllung cancer patients[J].Clinical Cancer Research,2009,15(6):2076-2084.
[27]Wang J,Ramakrishnan R,Tang Z,et al.QuantifyingEGFR alterations in the lung cancer genome withnanofluidic digital PCR arrays[J].Clin Chem,2010,56(4):623-632.
[28]Taly V,Pekin D,Benhaim L,et al.Multiplex picodr-oplet digital PCR to detect KRAS mutation in circulat-ing DNA from the plasma of colorectal cancer patients[J].Clin Chem,2013,59(12):1722-1731.
[29]Jacob H J.Next-generation sequencing for clinical di-agnostics[J].N Engl J Med,2013,369:1557-1558.
[30]White R A,Blainey P C,Fan H C,et al.Digital PCRprovides sensitive and absolute calibration for highthroughput sequencing[J].BMC Genomics,2009,10:116.
[31]Robin J D,Ludlow A T,LaRanger R,et al.Compari-son of DNA quantification methods for Next generationsequencing[J].Scientific Reports,2016,6:24067.
[32]Holst-Jensen A.Testing for genetically modified or-ganisms(GMOs):Past,present and future perspec-tives[J].Biotechnol Adv,2009,27:1071-1082.
[33]Settanni L,Corsetti A.The use of multiplex PCR todetect and differentiate food-and beverage-associatedmicroorganisms:A review[J].Journal of Microbiolog-ical Methods,2007,69:1-22.
[34]Cankar K,Stebih D,Dreo T,et al.Critical points ofDNA quantification by real-time PCR-effects of DNAextraction method and sample matrix on quantificationof genetically modified organisms[J].BMC Biotechnol,2006,6:37.
[35]Corbisier P,Bhat S,Partis L,et al.Absolute quantifi-cation of genetically modified MON810maize by digitalpolymerase chain reaction[J].Analytical and Bioana-lytical Chenistry,2010,396(6):2143-2150.
[36]Morisset D,Stebih D,Milavec M,et al.Quantitativeanalysis of food and feed samples with droplet digitalPCR[J/OL].PLoS One,2013,8(5):e62583.
[37]Fu W,Zhu P Y,Wang C G,et al.A highly sensitiveand specific method for the screening detection of ge-netically modified organisms based on digital PCR with-out pretreatment[J].Scientific Reports,2015,5:12715
重要提示:转载本站信息须注明来源:985论文网,具体权责及声明请参阅网站声明。
阅读提示:请自行判断信息的真实性及观点的正误,本站概不负责。
论文服务


 专科论文咨询
专科论文咨询
